Z-ДНК — одна из многих возможных структур двойной спирали ДНК, представляет собой левозакрученную двойную спираль (в отличие от правозакрученной, как наиболее распространённая форма В-ДНК[en]). Z-ДНК является одной из трёх биологически активных двойных спиральных структур ДНК, наряду с А-ДНК и В-ДНК, хотя точные её функции к настоящему моменту не определены[1].
История изучения
Левозакрученная ДНК впервые была открыта Робертом Уэллсом и коллегами при изучении полимера, образованного повторениями инозин-цитозина[2]. Они наблюдали «обратный» круговой дихроизм в таких ДНК, из чего сделали верный вывод, что её цепи обвивают друг друга в направлении налево. Впоследствии была опубликована кристаллическая структура Z-ДНК, где в ходе рентгеноструктурного анализа выяснилось, что она является первым однокристаллическим фрагментом ДНК (самокомплементарный гексамер ДНК d(CG)3). Было установлено, что Z-ДНК представляет собой левозакрученную двойную спираль ДНК из двух антипараллельных цепей, соединённых связями между парами азотистых оснований. Эти работы были проведены Эндрю Уонг (англ. Andrew Wang), Александром Ричем и их сотрудниками в Массачусетском технологическом институте[3].
В 1970 году было показано, что наиболее распространённая B-форма ДНК может переходить в Z-форму. В этом эксперименте было продемонстрировано, что круговой дихроизм полимера (dG-dC) в ультрафиолетовых лучах при в растворе 4М NaCl менялся на строго противоположный[4]. То, что при этом переходе В-форма перешла в Z-форму, было подтверждено результатами рамановской спектроскопии[5]. Кристаллизация соединения В- и Z-ДНК, проведённая в 2005 году[6], дала лучшее понимание потенциальной роли, которую Z-ДНК играет в клетке. Везде, где есть сегменты форм Z-ДНК, должны быть также В-Z-соединения на их концах, связывающие Z-форму с B-формой, встречающейся во всём остальном геноме.
В 2007 году была описана РНК-версия Z-ДНК как трансформированная форма двойной правозакрученной спирали A-РНК в левозакрученную спираль[7]. Переход от А-РНК в Z-РНК, тем не менее, был описан уже в 1984 году[8].
Структура
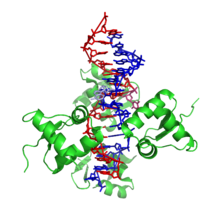
Z-ДНК значительно отличается от правозакрученных форм. Z-ДНК — левозакрученная и имеет первичную структуру, повторяющуюся через каждые 2 пары оснований. На один поворот спирали приходится 12 пар оснований. В отличие от А- и В-ДНК, в Z-ДНК большая бороздка слабо различима, малая бороздка узкая и глубокая[9]. Вообще, структура Z-ДНК энергетически невыгодна, хотя некоторые условия могут активизировать её формирования, как то: чередующиеся пуриново-пиримидиновые последовательности (особенно поли(dGC)2), негативная сверхспирализация ДНК, высокое содержание солей и некоторые катионы (все при физиологической температуре — 37 °C и pH 7,3—7,4). Z-ДНК может соединяться с B-ДНК в структуру, приводящую к вытеснению пар оснований (см. рис.)[10].

Ещё одной особенностью Z-ДНК является чередование конформаций нуклеотидных остатков. Дезоксицитидин находится в стандартной конформации: сахар в С2'-эндоконформации (см. рис.), а основание — в анти-конформации (то есть основание повёрнуто в сторону, противоположную гидроксильной группе при пятом атоме углерода; в таком положении находятся основания в полинуклеотидной цепи[11]). У дезоксигуанозина сахар находится в С3'-эндоконформации, а основание имеет крайне нетипичную син-конформацию[12].
Стэкинг оснований в Z-ДНК обладает новыми, присущими лишь этой форме свойствами. Так, стэкинговые взаимодействия имеются только между остатками цитозина противоположных цепей, а остатки гуанина вообще не взаимодействуют друг с другом[1].
Фосфаты в Z-ДНК не эквивалетны друг другу и удалены на различные расстояния от оси спирали; для гуаниновых нуклеотидов это расстояние равно 0,62 нм, а для цитозиновых — 0,76 нм. При этом соседние сахара «смотрят» в противоположные стороны, и из-за этого линия, последовательно соединяющая атомы фосфора в цепи, становится зигзагообразной (отсюда название — Z-ДНК)[1].
Структура Z-ДНК сложна для изучения, потому что она практически не существует в стабильной форме двойной спирали. Напротив, левозакрученная спираль Z-ДНК является временной структурой, появляющейся в результате биологической активности и быстро исчезающей[13].

Переход из В-ДНК в Z-ДНК
Как уже говорилось, В- и Z-формы способны переходить друг в друга. Это происходит при изменении ионной силы раствора или концентрации катионов, нейтрализующих отрицательный заряд фосфодиэфирного каркаса. При этом для перехода нет необходимости для расхождения цепей, он инициируется разрывом водородных связей у нескольких пар оснований, после чего гуанин фиксируется в син-конформации, водородные связи восстанавливаются, и основания вновь образуют уотсон-криковские пары. Область перехода движется по спирали в виде петли[1].
Предсказание структуры Z-ДНК
В настоящий момент возможно предсказать правдоподобную последовательность ДНК, находящейся в форме Z-ДНК. Алгоритм для предсказания склонности ДНК перестраиваться из В-формы в Z-форму, ZHunt, был написан в 1984 году д-ром P. Shing Ho из Массачусеткого технологического института[14]. Позже этот алгоритм был развит Трейси Кэмп и коллегами для определения мест образования Z-ДНК во всём геноме[15].
Алгоритм ZHunt доступен по ссылке Z-Hunt online.
Биологическое значение
Z-ДНК обнаружены у представителей всех трёх доменов жизни: архей (в частности, у галоархей[16]), бактерий и эукариот[9]. Пока чётких биологических функций Z-ДНК не определено, однако предположительно она участвует в регуляции экспрессии генов на уровне транскрипции. Действительно, достоверно известно, что с регуляцией экспрессии генов у эукариот связана последовательнось dm5-dG, которая в физиологических условиях находится в форме Z-ДНК. Эта регуляция может быть опосредована сверхспирализацией, связыванием с белками, специфическим к Z-ДНК, определёнными катионами типа спермидина[en] и метилированием дезоксицитидина[17].
Предположение о том, что Z-ДНК обеспечивает сверхспирализацию ДНК во время транскрипции[6][18], подтверждается тем, что потенциал к образованию Z-форм обнаруживается на участках, задействованных в активной транскрипции. Была показана связь мест образования Z-ДНК в генах 22-й хромосомы человека и известных для них сайтов начала транскрипции[15].
Z-ДНК образуется после начала транскрипции. Первый домен, связывающийся с Z-ДНК и имеющий к ней большое сродство, был обнаружен у фермента ADAR1[en] (РНК-специфической аденозиндеаминазы)[19][20] (этот домен получил название Z-альфа домена). Кристаллографические исследования и исследования, проведённые методом ядерного магнитного резонанса, подтвердили, что этот домен связывает Z-ДНК вне зависимости от её последовательности нуклеотидов[21][22][23]. Схожие участки были обнаружены в некоторых других белках, гомологичных ADAR1[20]. Идентификация Z-альфа домена легла в основу характеризации Z-РНК и соединения B- с Z-ДНК. Исследования показали, что домен ADAR1, связывающий Z-ДНК, позволяет этому ферменту локализоваться в местах активной транскрипции, где он и выполняет свою функцию — изменяет последовательность новообразованной РНК[24][25].
В 2003 году биофизик Александр Рич из Массачусетского технологического института заметил, что фактор вирулентности поксвируса, называемый E3L, имеет Z-альфа-родственный участок, схожий с белком млекопитающих, связывающим Z-ДНК[26][27]. В 2005 году Рич и коллеги выяснили, какое значение E3L имеет для поксвируса. При экспрессии генов E3L вызывает повышение транскрипции нескольких генов хозяйской клетки от 5 до 10 раз, причём эти гены блокируют способность клеток к саморазрушению (апоптозу) как к защитной реакции против инфекции.
Рич предположил, что Z-ДНК необходима для транскрипции и E3L стабилизирует Z-ДНК, таким образом увеличивая экспрессию антиапоптических генов. Он также выдвинул идею, что малые молекулы могут связываться с E3L, препятствуя соединению этого белка с Z-ДНК, и в итоге мешают экспрессии антиапоптозных генов. Потенциально это может быть использовано в основе метода защиты от оспы, вызываемой поксвирусами.
С помощью антител к Z-ДНК эта форма ДНК была обнаружена в междисковых областях политенных хромосом. Дело в том, что нуклеосомы имеются только у В-ДНК, а переход в Z-форму разрушает структуру нуклеосомы и, следовательно, состоящего из нуклеосом хроматина. В связи с этим предполагается, что Z-форма может выполнять какую-то регуляторную роль, тем более, переход В → Z обратим[1].
Установлено, что токсический эффект бромистого этидия на трипаносомы связан с переходом ихкинетопластной ДНК в Z-форму. Этот эффект обусловлен интеркаляцией[en] EtBr в ДНК, из-за чего ДНК теряет свою нативную структуру, расплетается, переходит в Z-форму и из-за этого становится неспособной к репликации[28].
Сравнение геометрических параметров некоторых форм ДНК
| Геометрический параметр | A-форма | B-форма | Z-форма |
|---|---|---|---|
| Направление | правозакрученная | правозакрученная | левозакрученная |
| Единица повтора | 1 пара оснований (п. о.) | 1 п. о. | 2 п. о. |
| Оборот (в градусах) | 32,7° | 35,9° | 60°/2 |
| Изгиб | 11 п. о. | 10,5 п. о. | 12 п. о. |
| Расположение п.о. относительно оси | +19° | −1.2° | −9° |
| Подъём вдоль оси | 2,3 Å (0,23 нм) | 3,32 Å (0,332 нм) | 3,8 Å (0,38 нм) |
| Наклон | 28,2 Å (2,82 нм) | 33,2 Å (3,32 нм) | 45,6 Å (4,56 нм) |
| Скрученность | +18° | +16° | 0° |
| Конформация основания | анти- | анти- | C: анти-, G: син- |
| Конформация сахара | C3'-эндо | C2'-эндо | C: C2'-эндо, G: C3'-эндо |
| Диаметр | 23 Å (2,3 нм) | 20 Å (2,0 нм) | 18 Å (1,8 нм) |
| Источники:[29][30][31] | |||

Примечания
- 1 2 3 4 5 Коничев, Севастьянова, 2012, с. 93.
- ↑ Mitsui; et al. (1970). “Physical and enzymatic studies on poly d(I-C)-poly d(I-C), an unusual double-helical DNA”. Nature (London). 228 (5277): 1166—1169. PMID 4321098.
- ↑ Wang AHJ, Quigley GJ, Kolpak FJ, Crawford JL, van Boom JH, Van der Marel G, Rich A (1979). “Molecular structure of a left-handed double helical DNA fragment at atomic resolution”. Nature (London). 282 (5740): 680—686. Bibcode:1979Natur.282..680W. DOI:10.1038/282680a0. PMID 514347.
- ↑ Pohl FM, Jovin TM (1972). “Salt-induced co-operative conformational change of a synthetic DNA: equilibrium and kinetic studies with poly(dG-dC)”. J. Mol. Biol. 67: 375–396. DOI:10.1016/0022-2836(72)90457-3. PMID 5045303.
- ↑ Thamann TJ, Lord RC, Wang AHJ, Rich A (1981). “High salt form of poly(dG-dC)•poly(dG-dC) is left handed Z-DNA: raman spectra of crystals and solutions”. Nucl. Acids Res. 9: 5443—5457. DOI:10.1093/nar/9.20.5443. PMID 7301594.
- 1 2 Ha SC, Lowenhaupt K, Rich A, Kim YG, Kim KK (2005). “Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases”. Nature. 437 (7062): 1183—1186. Bibcode:2005Natur.437.1183H. DOI:10.1038/nature04088. PMID 16237447.
- ↑ Placido D, Brown BA 2nd, Lowenhaupt K, Rich A, Athanasiadis A (2007). “A left-handed RNA double helix bound by the Zalpha domain of the RNA-editing enzyme ADAR1”. Structure. 15 (4): 395—404. DOI:10.1016/j.str.2007.03.001. PMC 2082211. PMID 17437712.
- ↑ Hall K, Cruz P, Tinoco I Jr, Jovin TM, van de Sande JH (October 1984). “'Z-RNA'--a left-handed RNA double helix”. Nature. 311 (5986): 584—586. Bibcode:1984Natur.311..584H. DOI:10.1038/311584a0. PMID 6482970. Используется устаревший параметр
|month=(справка) - 1 2 Nelson, Cox, 2008, p. 281.
- ↑ de Rosa M, de Sanctis D, Rosario AL, Archer M, Rich A, Athanasiadis A, Carrondo MA (2010-05-18). “Crystal structure of a junction between two Z-DNA helices”. Proc Natl Acad Sci USA. 107 (20): 9088—9092. Bibcode:2010PNAS..107.9088D. DOI:10.1073/pnas.1003182107. PMC 2889044. PMID 20439751.
- ↑ Коничев, Севастьянова, 2012, с. 82.
- ↑ Коничев, Севастьянова, 2012, с. 92.
- ↑ Zhang H, Yu H, Ren J, Qu X (2006). “Reversible B/Z-DNA transition under the low salt condition and non-B-form polydApolydT selectivity by a cubane-like europium-L-aspartic acid complex”. Biophysical Journal. 90 (9): 3203—3207. Bibcode:2006BpJ....90.3203Z. DOI:10.1529/biophysj.105.078402. PMC 1432110. PMID 16473901. Архивировано из оригинала 2008-10-12. Проверено 2013-02-09. Используется устаревший параметр
|deadlink=(справка) - ↑ Ho PS, Ellison MJ, Quigley GJ, Rich A (1986). “A computer aided thermodynamic approach for predicting the formation of Z-DNA in naturally occurring sequences”. EMBO Journal. 5 (10): 2737—2744. PMC 1167176. PMID 3780676.
- 1 2 Champ PC, Maurice S, Vargason JM, Camp T, Ho PS (2004). “Distributions of Z-DNA and nuclear factor I in human chromosome 22: a model for coupled transcriptional regulation”. Nucleic Acids Res. 32 (22): 6501—6510. DOI:10.1093/nar/gkh988. PMC 545456. PMID 15598822.
- ↑ Paul Blum. Archaea: Ancient Microbes, Extreme Environments, and the Origin of Life. — Academic Press, 2001. — Vol. 50. — P. 206. — (Advances in Applied Microbiology).
- ↑ Коничев, Севастьянова, 2012, с. 93—94.
- ↑ {{cite journal − |author=Rich A, Zhang S |year=2003 − |title=Timeline: Z-DNA: the long road to biological function − |journal=Nature Review Genetics |volume=4 |issue=7 |pages=566–572 − |pmid=12838348 |doi=10.1038/nrg1115 − }}
- ↑ Herbert A, Rich A (1993). “A method to identify and characterize Z-DNA binding proteins using a linear oligodeoxynucleotide”. Nucleic Acids Res. 21 (11): 2669—72. DOI:10.1093/nar/21.11.2669. PMC 309597. PMID 8332463.
- 1 2 Herbert A, Alfken J, Kim YG, Mian IS, Nishikura K, Rich A (1997). “A Z-DNA binding domain present in the human editing enzyme, double-stranded RNA adenosine deaminase”. Proc Natl Acad Sci USA. 94 (16): 8421—6. Bibcode:1997PNAS...94.8421H. DOI:10.1073/pnas.94.16.8421. PMC 22942. PMID 9237992.
- ↑ Herbert A, Schade M, Lowenhaupt K, Alfken J, Schwartz T, Shlyakhtenko LS, Lyubchenko YL, Rich A (1998). “The Zalpha domain from human ADAR1 binds to the Z-DNA conformer of many different sequences”. Nucleic Acids Res. 26 (15): 2669—72. DOI:10.1093/nar/26.15.3486. PMC 147729. PMID 9671809.
- ↑ Schwartz T, Rould MA, Lowenhaupt K, Herbert A, Rich A (1999). “Crystal structure of the Zalpha domain of the human editing enzyme ADAR1 bound to left-handed Z-DNA”. Science. 284 (5421): 1841—5. DOI:10.1126/science.284.5421.1841. PMID 10364558.
- ↑ Schade M, Turner CJ, Kühne R, Schmieder P, Lowenhaupt K, Herbert A, Rich A, Oschkinat H (1999). “The solution structure of the Zalpha domain of the human RNA editing enzyme ADAR1 reveals a prepositioned binding surface for Z-DNA”. Proc Natl Acad Sci USA. 96 (22): 2465—70. Bibcode:1999PNAS...9612465S. DOI:10.1073/pnas.96.22.12465. PMC 22950. PMID 10535945.
- ↑ Herbert A, Rich A (2001). “The role of binding domains for dsRNA and Z-DNA in the in vivo editing of minimal substrates by ADAR1”. Proc Natl Acad Sci USA. 98 (21): 12132—7. Bibcode:2001PNAS...9812132H. DOI:10.1073/pnas.211419898. PMID 11593027.
- ↑ Halber D. Scientists observe biological activities of 'left-handed' DNA. MIT News Office (11 сентября 1999). Проверено 29 сентября 2008. Архивировано 16 февраля 2013 года.
- ↑ Kim YG, Muralinath M, Brandt T, Pearcy M, Hauns K, Lowenhaupt K, Jacobs BL, Rich A (2003). “A role for Z-DNA binding in vaccinia virus pathogenesis”. Proc Natl Acad Sci USA. 100 (12): 6974—6979. Bibcode:2003PNAS..100.6974K. DOI:10.1073/pnas.0431131100. PMC 165815. PMID 12777633.
- ↑ Kim YG, Lowenhaupt K, Oh DB, Kim KK, Rich A (2004). “Evidence that vaccinia virulence factor E3L binds to Z-DNA in vivo: Implications for development of a therapy for poxvirus infection”. Proc Natl Acad Sci USA. 101 (6): 1514—1518. Bibcode:2004PNAS..101.1514K. DOI:10.1073/pnas.0308260100. PMC 341766. PMID 14757814.
- ↑ Roy Chowdhury, Arnab; Bakshi, Rahul; Wang, Jianyang; Yildirir, Gokben; Liu, Beiyu; Pappas-Brown, Valeria; Tolun, Gökhan; Griffith, Jack D.; Shapiro, Theresa A.; Jensen, Robert E.; Englund, Paul T.; Ullu, Elisabetta (16 December 2010). “The Killing of African Trypanosomes by Ethidium Bromide”. PLoS Pathogens. 6 (12): e1001226. DOI:10.1371/journal.ppat.1001226. Используется устаревший параметр
|coauthors=(справка) - ↑ Sinden, Richard R. DNA structure and function. — 1st. — Academic Press, 1994-01-15. — P. 398. — ISBN 0-126-45750-6.
- ↑ Rich A, Norheim A, Wang AHJ (1984). “The chemistry and biology of left-handed Z-DNA”. Annual Review of Biochemistry. 53 (1): 791—846. DOI:10.1146/annurev.bi.53.070184.004043. PMID 6383204.
- ↑ Ho PS (1994-09-27). “The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA”. Proc Natl Acad Sci USA. 91 (20): 9549—9553. Bibcode:1994PNAS...91.9549H. DOI:10.1073/pnas.91.20.9549. PMC 44850. PMID 7937803.
Литература
- Коничев А. С., Севастьянова Г. А. Молекулярная биология. — Издательский центр «Академия», 2012. — 400 с. — ISBN 978-5-7695-9147-1.
- David L. Nelson, Michael M. Cox. Lehninger Principles of biochemistry. — Fifth edition. — New York: W. H. Freeman and company, 2008. — 1158 p. — ISBN 978-0-7167-7108-1.
Эта статья входит в число хороших статей русскоязычного раздела Википедии. |
Данная страница на сайте WikiSort.ru содержит текст со страницы сайта "Википедия".
Если Вы хотите её отредактировать, то можете сделать это на странице редактирования в Википедии.
Если сделанные Вами правки не будут кем-нибудь удалены, то через несколько дней они появятся на сайте WikiSort.ru .