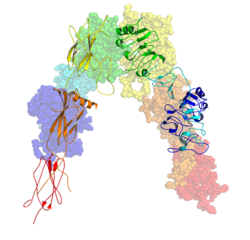
Инсулиновый рецептор (IR) (англ. insulin receptor) — трансмембранный рецептор, который активируется инсулином, IGF-I, IGF-II и принадлежит к большому классу тирозинкиназных рецепторов[1]. Инсулиновый рецептор играет ключевую роль в регуляции гомеостаза глюкозы, функционального процесса, который при дегенеративных условиях может привести к ряду клинических проявлений, в том числе диабету и раку[2][3]. Биохимически инсулиновый рецептор кодируется одним геном INSR, альтернативный сплайсинг которого в ходе транскрипции производит либо к IR-A-либо к IR-B-изоформам[4]. Последующие посттрансляционные события каждой изоформы приводят к формированию протеолитически расщеплённых α и β-субъединиц, которые при объединении, в конечном счете способны к димеризации с получением дисульфидно связанного трансмембранного инсулинового рецептора массой ≈ 320 кДа[4].
Структура
Первоначально транскрипты альтернативных вариантов сплайсинга гена INSR транслируются с образованием одного из двух мономерных изомеров: IR-A, в котором вырезан экзон 11, и IR-B, в котором есть экзон 11. Включение экзона 11 приводит к добавлению 12 аминокислот выше фурина в сайте протеолитического расщепления.

При димеризации рецептора, после протеолитического расщепления α- и β-цепей, дополнительные 12 аминокислот остаются на С-конце α-цепи (обозначенной αCT), где они предположительно влияют на взаимодействия рецептора и лиганда[5].
Каждый изомерический мономер структурно разбит на 8 различных доменов; домен лейцин-обогащённых повторов (L1, остатки 1-157), регион, богатый цистеином (CR, остатки 158—310), дополнительный домен лейцин-обогащённых повторов (L2, остатки 311—470), три типа доменов фибронектина III; FnIII-1 (остатки 471—595), FnIII-2 (остатки 596—808) и FnIII-3 (остатки 809—906). Кроме того, вставной домен (ID, остатки 638—756), находящийся в пределах FnIII-2, содержащий сайт расщепления α/β фурина, протеолиз которого действуют как в IDα- так и IDβ-доменах. В β-цепи ниже области FnIII-3 находится трансмембранная спираль и внутриклеточная околомембранная область, непосредственно выше внутриклеточного каталитического тирозинкиназного домена, ответственного за активацию внутриклеточных сигнальных путей[6]. При расщеплении мономера на соответствующие α- и β-цепи рецептор гомо- или гетеродимеризуется через ковалентно дисульфидную связь, а между мономерами в димере образуется две дисульфидные связи, идущие от каждого α-цепи. Общая структура 3D эктодомена[en], обладает четырьмя сайтами связывания лиганда, напоминает перевернутую V. Каждый мономер поворачивается примерно 2 раза вокруг оси, проходящей параллельно перевернутой V, L2 и FnIII-1 доменам от каждого мономера, формирующего вершину перевернутой V[6][7].
Связывание лиганда
Эндогенные лиганды инсулинового рецептора включают инсулин, IGF-I и IGF-II. Связывание лиганда с α-цепями эктодомена IR вызывает структурные изменения в рецепторе, ведущие к автофосфорилированию различных остатков тирозина во внутриклеточном домене TK в β-цепи. Эти изменения способствовуют рекрутированию определенных адаптерных белков[en], таких как белки субстрата инсулинового рецептора (IRS) в дополнение к SH2-B[en] (гомолог Src 2 — B), APS и протеинфосфатазы, таких как PTP1B[en], в конечном итоге, способствующих последующим процессам, связанным с гомеостазом глюкозы в крови[8].

Строго говоря, отношения между инсулиновым рецептором и лигандом показывают сложные аллостерические свойства. На это указывает график Скэтчарда[en] который показывает, что измеренное соотношение инсулинового рецептора, связанного с лигандом, относительно несвязанного лиганда не следует линейной зависимости относительно изменений концентрации инсулинового рецептора, связанного с лигандом, давая основание предположить, что инсулиновый рецептор и его лиганд взаимодействуют по механизму кооперативно связаны[en][9]. Кроме того, наблюдение, что скорость диссоциации IR-лиганда увеличивается при добавлении несвязанного лиганда предполагает, что природа этого сотрудничества отрицательна; иначе говоря, начальное связывание лиганда с IR ингибирует дополнительное связывание со своим вторым активным сайтом, демонстрируя аллостерическое ингибирование[9].
Хотя точный механизм связывания IR с его лигандом структурно ещё не выяснен, с точки зрения системной биологии, биологически значимое предсказание кинетики[en] IR-лиганд (инсулин/IGF-I) было определено в контексте доступной в настоящий момент структуры эктодомена IR[6][7].
Эти модели утверждают, что каждый мономер IR имеет 2 инсулиновых сайта связывания; Сайт 1, который связывается с «классической» поверхностью связывания инсулина: состоящей из L1 плюс αCT доменов и сайта 2, состоящий из петель на стыке FnIII-1 и FnIII-2, по прогнозам, связывающихся с «новым» гексамерным лицом сайта связывания инсулина[1]. Так как каждый мономер предоставляет IR эктодомену представление 3D «зеркальной» взаимодополняемости, N-терминальный сайт 1 из одного мономера, в конечном счете сталкивается с C-терминальным сайтом 2 второго мономера, что также верно для каждого зеркального дополнения мономеров (противоположная сторона структуры эктодомена). Текущая литература отличает сайты связывания дополнений, назначив на сайте 1 и 2 мономерные сайты дополнений, как 3 и 4 или как сайт 1' и 2' соответственно[1][10].
Таким образом, эти модели утверждают, что каждый IR может связываться с молекулой инсулина (который имеет две связывающих поверхности) в 4 местах, посредством сайтов 1, 2, (3/1') или (4/2'). Поскольку каждый сайт 1 проксимально сталкивается с сайтом 2, по прогнозам, произойдет связывания инсулина конкретным сайтом, «сшивание»[en] с помощью лиганда между мономерами, (то есть [мономер 1 Сайт 1 — Инсулин — мономер 2 сайт (4/2')] или [мономер 1 сайт 2 — Инсулин — мономер 2-сайт (3/1')]). В соответствии с действующим математическим моделированием IR-инсулиновой кинетики, есть два важных последствия для событий сшивания инсулина; 1. в вышеупомянутом наблюдении отрицательное взаимодействие IR и его лиганда, после связывания лиганда с IR снижается и 2. физическое воздействие приводит к сшиванию эктодомена в такой конформации, которая необходима для наступления событий внутриклеточного фосфорилирования тирозина (то есть эти события служат требованием к активации рецептора с последующим поддержанием гомеостаза глюкозы в крови)[8].
Биологическое значение
Рецепторы тирозинкиназы[en], в том числе инсулиновый рецептор, опосредуют свою активность, вызывая добавление фосфатной группы к конкретным тирозинам в клетках определенных белков. В «подложке» белки, которые фосфорилируются инсулиновым рецептором включает белок, называемый «IRS-1» для «инсулинового рецептора подложки 1». Связывания и фосфорилирования IRS-1 в конечном итоге приводит к увеличению высокого сродства молекул транспортёра глюкозы (GLUT4) на внешней мембране инсулиночувствительных тканей, в том числе мышечных клеток и жировой ткани, и, следовательно, к увеличению поглощения глюкозы из крови в этих тканях. Другими словами, глюкозный транспортёр GLUT4 транспортируется из клеточных везикул к клеточной поверхности, где он затем может опосредовать транспорт глюкозы в клетку.

Патология
Основная деятельность активации инсулинового рецептора — индуцировать поглощение глюкозы. По этой причине «нечувствительность инсулина», или снижение сигнализации инсулинового рецептора, приводит к сахарному диабету 2 типа — клетки неспособны принять глюкозу и в результате — гипергликемия (повышение циркуляции глюкозы) и все последствия диабета.
Пациенты с инсулинорезистентностью могут иметь признаки чёрного акантоза.
Несколько пациентов с гомозиготной мутацией гена INSR были описаны, как получившие синдром Донохью[en]. Это аутосомно-рецессивные нарушения делают совершенно нефункциональными инсулиновые рецепторы. Эти пациенты имеют низкорасположенные, часто выпуклые, уши, ноздри, утолщенные губы и сильную задержку роста. В большинстве случаев, прогноз для этих пациентов является крайне бедным, смертельный исход случается в течение первого года жизни. Другие мутации того же гена вызывают менее тяжелый синдром Робсона-Менденхолла[en], при которых пациенты имеют характерно аномальные зубы, гипертрофированные дёсны и расширение шишковидной железы. Оба заболевания представляют флуктацию уровня глюкозы: после приема пищи глюкоза изначально очень высока, а затем резко падает до аномально низких уровней[11].
Регуляция экспрессии генов
Активированные IRS-1 действуют в качестве вторичного мессенджера в клетке, чтобы стимулировать транскрипцию инсулинорегулируемых генов. Во-первых, белок Grb2 связывает Р-Tyr остаток IRS-1 в его домене SH2[en]. Grb2 становится в состоянии связать SOS, который в свою очередь катализирует замену связанного GDP с GTP в Ras, G-белка. Этот белок затем начинает каскад фосфорилирования, что приводит к активации митогеноактивируемой протеинкиназы (МАРК), которая входит в ядро и фосфорилирует различные факторы ядерной транскрипции (например, Elk1).
Стимуляция синтеза гликогена
Синтез гликогена также стимулируется инсулиновым рецептором с помощью IRS-1. В этом случае это SH2-домен[en] из киназы PI-3 (PI-3K[en]), который связывает P-Tyr из IRS-1. Теперь активации PI-3K может конвертировать мембранные липидные фосфатидилинозитол 4,5-бисфосфата[en] (PIP2) до фосфатидилинозит 3,4,5-трифосфата[en] (PIP3). Это косвенно активирует протеинкиназу PKB (Akt) с помощью фосфорилирования. РКВ затем фосфорилирует несколько целевых белков, в том числе киназу гликогенсинтазы 3[en] (GSK-3). GSK-3 несёт ответственность за фосфорилирование (и, следовательно, деактивацию) гликогенсинтазы. Когда GSK-3 фосфорилируется, он отключается, и предотвращается деактивации гликогенсинтазы. Этим окольным путём инсулин увеличивает синтез гликогена.
Деградация инсулина
После того как молекула инсулина стыкуется с рецептором и активирует его, она может быть выпущена обратно во внеклеточную среду, или может быть деградирована в клетке. Деградация обычно включает эндоцитоз инсулино-рецепторного комплекса с последующим действием фермента, разрушающего инсулин. Большинство молекул инсулина деградируют в клетках печени. Было подсчитано, что типичная молекула инсулина деградирует приблизительно через 71 минуту после первоначального выпуска в кровоток[12].
Взаимодействия
Инсулиновый рецептор, как было выявлено, взаимодействует с ENPP1[13], PTPN11[14][15], GRB10[16][17][18][19][20], GRB7[21], PRKCD[22][23], IRS1[24][25], SH2B1[26][27] и MAD2L1[28].
Примечания
- 1 2 3 Ward CW, Lawrence MC (April 2009). “Ligand-induced activation of the insulin receptor: a multi-step process involving structural changes in both the ligand and the receptor”. BioEssays. 31 (4): 422—34. DOI:10.1002/bies.200800210. PMID 19274663.
- ↑ Ebina Y, Ellis L (April 1985). “The human insulin receptor cDNA: the structural basis for hormone-activated transmembrane signalling”. Cell. 40 (4): 747—58. DOI:10.1016/0092-8674(85)90334-4. PMID 2859121.
- ↑ Malaguarnera R, Belfiore A (February 2012). “Proinsulin Binds with High Affinity the Insulin Receptor Isoform A and Predominantly Activates the Mitogenic Pathway”. Endocrinology. Epub (5): 2152—63. DOI:10.1210/en.2011-1843. PMID 22355074.
- 1 2 Belfiore A, Frasca F (Oct 2009). “Insulin receptor isoforms and insulin receptor/insulin-like growth factor receptor hybrids in physiology and disease”. Endocr Rev. 30 (6): 586—623. DOI:10.1210/er.2008-0047. PMID 19752219.
- ↑ Knudsen L, De Meyts P, Kiselyov VV. (Feb 2012). “Insight into the molecular basis for the kinetic differences between the two insulin receptor isoforms”. Biochemical J. 440 (3): 397—403. DOI:10.1042/BJ20110550. PMID 21838706.
- 1 2 3 Smith BJ, Huang K (April 2010). “Structural resolution of a tandem hormone-binding element in the insulin receptor and its implications for design of peptide agonists”. PNAS. 107 (15): 6771—6. Bibcode:2010PNAS..107.6771S. DOI:10.1073/pnas.1001813107. PMC 2872410. PMID 20348418.
- 1 2 McKern NM, Lawrence MC, Ward CW; et al. (Sep 2006). “Structure of the insulin receptor ectodomain reveals a folded-over conformation”. Nature. 7108 (443): 218—21. Bibcode:2006Natur.443..218M. DOI:10.1038/nature05106. PMID 16957736.
- 1 2 Kiselyov VV, Versteyhe S, Gauguin L, De Meyts P (Feb 2009). “Harmonic oscillator model of the insulin and IGF1 receptors' allosteric binding and activation”. Mol Syst Biol. 253 (5). DOI:10.1038/msb.2008.78. PMC 2657531. PMID 19225456.
- 1 2 de Meyts P, Roth J, Neville DM Jr, Gavin JR 3rd, Lesniak MA (Nov 1973). “Insulin interactions with its receptors: experimental evidence for negative cooperativity”. Biochem Biophys Res Commun. 55 (1): 154—61. DOI:10.1016/S0006-291X(73)80072-5. PMID 4361269.
- ↑ Kiselyov VV, Versteyhe S, Gauguin L, De Meyts P (Feb 2009). “Harmonic oscillator model of the insulin and IGF1 receptors' allosteric binding and activation”. Mol Syst Biol. 253 (5). DOI:10.1038/msb.2008.78. PMC 2657531. PMID 19225456.
- ↑ Longo N, Wang Y, Smith SA, Langley SD, DiMeglio LA, Giannella-Neto D (2002). “Genotype-phenotype correlation in inherited severe insulin resistance”. Hum. Mol. Genet. 11 (12): 1465—75. DOI:10.1093/hmg/11.12.1465. PMID 12023989.
- ↑ Duckworth WC, Bennett RG, Hamel FG (1998). “Insulin degradation: progress and potential”. Endocr. Rev. 19 (5): 608—24. DOI:10.1210/er.19.5.608. PMID 9793760.
- ↑ Maddux, B A; Goldfine I D (Jan 2000). “Membrane glycoprotein PC-1 inhibition of insulin receptor function occurs via direct interaction with the receptor alpha-subunit”. Diabetes. UNITED STATES. 49 (1): 13—9. DOI:10.2337/diabetes.49.1.13. ISSN 0012-1797. PMID 10615944.
- ↑ Maegawa, H; Ugi S; Adachi M; Hinoda Y; Kikkawa R; Yachi A; Shigeta Y; Kashiwagi A (Mar 1994). “Insulin receptor kinase phosphorylates protein tyrosine phosphatase containing Src homology 2 regions and modulates its PTPase activity in vitro”. Biochem. Biophys. Res. Commun. UNITED STATES. 199 (2): 780—5. DOI:10.1006/bbrc.1994.1297. ISSN 0006-291X. PMID 8135823.
- ↑ Kharitonenkov, A; Schnekenburger J; Chen Z; Knyazev P; Ali S; Zwick E; White M; Ullrich A (Dec 1995). “Adapter function of protein-tyrosine phosphatase 1D in insulin receptor/insulin receptor substrate-1 interaction”. J. Biol. Chem. UNITED STATES. 270 (49): 29189—93. DOI:10.1074/jbc.270.49.29189. ISSN 0021-9258. PMID 7493946.
- ↑ Langlais, P; Dong L Q; Hu D; Liu F (Jun 2000). “Identification of Grb10 as a direct substrate for members of the Src tyrosine kinase family”. Oncogene. ENGLAND. 19 (25): 2895—903. DOI:10.1038/sj.onc.1203616. ISSN 0950-9232. PMID 10871840.
- ↑ Hansen, H; Svensson U; Zhu J; Laviola L; Giorgino F; Wolf G; Smith R J; Riedel H (Apr 1996). “Interaction between the Grb10 SH2 domain and the insulin receptor carboxyl terminus”. J. Biol. Chem. UNITED STATES. 271 (15): 8882—6. DOI:10.1074/jbc.271.15.8882. ISSN 0021-9258. PMID 8621530.
- ↑ Liu, F; Roth R A (Oct 1995). “Grb-IR: a SH2-domain-containing protein that binds to the insulin receptor and inhibits its function”. Proc. Natl. Acad. Sci. U.S.A. UNITED STATES. 92 (22): 10287—91. Bibcode:1995PNAS...9210287L. DOI:10.1073/pnas.92.22.10287. ISSN 0027-8424. PMC 40781. PMID 7479769.
- ↑ He, W; Rose D W; Olefsky J M; Gustafson T A (Mar 1998). “Grb10 interacts differentially with the insulin receptor, insulin-like growth factor I receptor, and epidermal growth factor receptor via the Grb10 Src homology 2 (SH2) domain and a second novel domain located between the pleckstrin homology and SH2 domains”. J. Biol. Chem. UNITED STATES. 273 (12): 6860—7. DOI:10.1074/jbc.273.12.6860. ISSN 0021-9258. PMID 9506989.
- ↑ Frantz, J D; Giorgetti-Peraldi S; Ottinger E A; Shoelson S E (Jan 1997). “Human GRB-IRbeta/GRB10. Splice variants of an insulin and growth factor receptor-binding protein with PH and SH2 domains”. J. Biol. Chem. UNITED STATES. 272 (5): 2659—67. DOI:10.1074/jbc.272.5.2659. ISSN 0021-9258. PMID 9006901.
- ↑ Kasus-Jacobi, A; Béréziat V; Perdereau D; Girard J; Burnol A F (Apr 2000). “Evidence for an interaction between the insulin receptor and Grb7. A role for two of its binding domains, PIR and SH2”. Oncogene. ENGLAND. 19 (16): 2052—9. DOI:10.1038/sj.onc.1203469. ISSN 0950-9232. PMID 10803466.
- ↑ Braiman, L; Alt A; Kuroki T; Ohba M; Bak A; Tennenbaum T; Sampson S R (Apr 2001). “Insulin induces specific interaction between insulin receptor and protein kinase C delta in primary cultured skeletal muscle”. Mol. Endocrinol. United States. 15 (4): 565—74. DOI:10.1210/mend.15.4.0612. ISSN 0888-8809. PMID 11266508.
- ↑ Rosenzweig, Tovit; Braiman Liora; Bak Asia; Alt Addy; Kuroki Toshio; Sampson Sanford R (Jun 2002). “Differential effects of tumor necrosis factor-alpha on protein kinase C isoforms alpha and delta mediate inhibition of insulin receptor signaling”. Diabetes. United States. 51 (6): 1921—30. DOI:10.2337/diabetes.51.6.1921. ISSN 0012-1797. PMID 12031982.
- ↑ Aguirre, Vincent; Werner Eric D; Giraud Jodel; Lee Yong Hee; Shoelson Steve E; White Morris F (Jan 2002). “Phosphorylation of Ser307 in insulin receptor substrate-1 blocks interactions with the insulin receptor and inhibits insulin action”. J. Biol. Chem. United States. 277 (2): 1531—7. DOI:10.1074/jbc.M101521200. ISSN 0021-9258. PMID 11606564.
- ↑ Sawka-Verhelle, D; Tartare-Deckert S, White M F, Van Obberghen E (Mar 1996). “Insulin receptor substrate-2 binds to the insulin receptor through its phosphotyrosine-binding domain and through a newly identified domain comprising amino acids 591–786”. J. Biol. Chem. UNITED STATES. 271 (11): 5980—3. DOI:10.1074/jbc.271.11.5980. ISSN 0021-9258. PMID 8626379. Используется устаревший параметр
|coauthors=(справка) - ↑ Kotani, K; Wilden P; Pillay T S (Oct 1998). “SH2-Balpha is an insulin-receptor adapter protein and substrate that interacts with the activation loop of the insulin-receptor kinase”. Biochem. J. ENGLAND. 335 (1): 103—9. ISSN 0264-6021. PMC 1219757. PMID 9742218.
- ↑ Nelms, K; O'Neill T J; Li S; Hubbard S R; Gustafson T A; Paul W E (Dec 1999). “Alternative splicing, gene localization, and binding of SH2-B to the insulin receptor kinase domain”. Mamm. Genome. UNITED STATES. 10 (12): 1160—7. DOI:10.1007/s003359901183. ISSN 0938-8990. PMID 10594240.
- ↑ O'Neill, T J; Zhu Y; Gustafson T A (Apr 1997). “Interaction of MAD2 with the carboxyl terminus of the insulin receptor but not with the IGFIR. Evidence for release from the insulin receptor after activation”. J. Biol. Chem. UNITED STATES. 272 (15): 10035—40. DOI:10.1074/jbc.272.15.10035. ISSN 0021-9258. PMID 9092546.
Литература
- Pearson RB, Kemp BE (1991). “Protein kinase phosphorylation site sequences and consensus specificity motifs: tabulations”. Meth. Enzymol. 200: 62—81. DOI:10.1016/0076-6879(91)00127-I. PMID 1956339.
- Joost HG (1995). “Structural and functional heterogeneity of insulin receptors”. Cell. Signal. 7 (2): 85—91. DOI:10.1016/0898-6568(94)00071-I. PMID 7794689.
- O'Dell SD, Day IN (1998). “Insulin-like growth factor II (IGF-II)”. Int. J. Biochem. Cell Biol. 30 (7): 767—71. DOI:10.1016/S1357-2725(98)00048-X. PMID 9722981.
- Lopaczynski W (1999). “Differential regulation of signaling pathways for insulin and insulin-like growth factor I”. Acta Biochim. Pol. 46 (1): 51—60. PMID 10453981.
- Sasaoka T, Kobayashi M (2000). “The functional significance of Shc in insulin signaling as a substrate of the insulin receptor”. Endocr. J. 47 (4): 373—81. DOI:10.1507/endocrj.47.373. PMID 11075717.
- Perz M, Torlińska T (2001). “Insulin receptor—structural and functional characteristics”. Med. Sci. Monit. 7 (1): 169—77. PMID 11208515.
- Benaim G, Villalobo A (2002). “Phosphorylation of calmodulin. Functional implications”. Eur. J. Biochem. 269 (15): 3619—31. DOI:10.1046/j.1432-1033.2002.03038.x. PMID 12153558.
Данная страница на сайте WikiSort.ru содержит текст со страницы сайта "Википедия".
Если Вы хотите её отредактировать, то можете сделать это на странице редактирования в Википедии.
Если сделанные Вами правки не будут кем-нибудь удалены, то через несколько дней они появятся на сайте WikiSort.ru .