| Ротавирусы | |||||||||
 Компьютерная реконструкция ротавируса, основанная на нескольких микрографах | |||||||||
| Научная классификация | |||||||||
|---|---|---|---|---|---|---|---|---|---|
|
промежуточные ранги
| |||||||||
| Международное научное название | |||||||||
|
Rotavirus | |||||||||
| Виды[2] | |||||||||
| |||||||||
| Группа по Балтимору | |||||||||
|
III: дцРНК-вирусы | |||||||||
| |||||||||
Ротавирусы[3] (лат. Rotavirus) — род вирусов с двунитевой сегментированной РНК, принадлежащий семейству реовирусов (Reoviridae), возбудители ротавирусной инфекции.
Внешний вид частиц напоминает «колесо с широкой ступицей, короткими спицами и чётко очерченным ободком»[4], из-за чего они и получили своё название (лат. rota — «колесо»).
Виды
Известно 9 видов данного рода, обозначаемых латинскими буквами A—I[2]. Человек может инфицироваться видами A, B и C, при этом возбудителем более 90 % ротавирусных инфекций является наиболее часто встречаемый вид — ротавирус A. Виды с A по E могут вызывать болезни у других животных[5]. К виду Ротавирус A относится несколько серотипов[6]. Как и в случае с вирусом гриппа, здесь применяется двойная классификация по подтипам поверхностных белков: серотипы G определяются вариациями гликопротеина VP7, а серотипы P — протеазочувствительным белком VP4[7]. Поскольку гены, определяющие G- и P-типы, наследуются независимо друг от друга, встречаются различные их комбинации[8].
Структура
Геном ротавируса состоит из 11 уникальных двунитевых молекул РНК, состоящих в общей сложности из 18 555 нуклеотидов. Нити нумеруются от 1 до 11 в порядке уменьшения длины, каждая представляет собой один ген. Каждый ген кодирует один белок, за исключением гена 9, кодирующего два белка[9]. РНК окружена трёхслойным белковым капсидом в форме усечённого икосаэдра. Каждый из слоёв сложен отдельным вирусным белком. Внутренний и средний слои перфорированы каналами. Средний слой визуально содержит «спицы колеса» (белок VP6) и является важнейшим компонентом вириона.[10] Размер вируса — 76,5 нм в диаметре[11][12], суперкапсида нет[5].
Белки
Вирион сформирован шестью структурными вирусными протеинами (VP), которые обозначаются как VP1, VP2, VP3, VP4, VP6 и VP7. Инфицированная вирусом клетка продуцирует также шесть неструктурных белков (NSP), не являющихся частью вирусной частицы. Они обозначаются: NSP1, NSP2, NSP3, NSP4, NSP5 и NSP6.
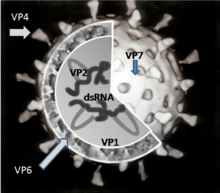
По крайней мере шесть из двенадцати кодируемых геномом вируса белков связываются с РНК[13]. Роль этих белков в репликации вируса до конца не выяснена; их функции, как считается, относятся к синтезу РНК и его упаковке в вирион, транспортировке мРНК к месту репликации, трансляции мРНК и регуляции экспрессии генов[14].
Структурные белки
VP1 располагается в ядре вирусной частицы и представляет собой фермент — РНК-полимеразу[15]. В инфицированной клетке фермент осуществляет синтез мРНК для дальнейшего производства вирусных белков, а также синтез сегментов РНК вирусного генома для новых вирионов.
VP2 формирует ядро вируса и связывает вирусный геном[16].
VP3 также составляет ядро вириона и является ферментом гуанилил-трансферазой. Данный фермент катализирует образование 5'-кэп во время процессинга пре-мРНК[17]. Кэп стабилизирует вирусную мРНК, защищая её от утилизации нуклеазами[18].
VP4 расположен на поверхности вириона и выступает с неё в виде шипа[19]. Он связывается с рецепторами на поверхности клеток и управляет внедрением вируса в клетку[20]. Для того, чтобы вирус смог вызывать инфекцию, VP4 должен быть модифицирован находящимся в кишечнике ферментом трипсином в VP5* и VP8*[21]. Именно VP4 определяет вирулентность вируса. VP4 используется для серотипической классификации ротавирусов наряду с VP7.
VP6 формирует толщу капсида. Этот белок крайне антигенен и может использоваться для определения вида ротавируса[22]. Этот белок используется для определения инфекции, вызванной ротавирусом А, в лабораторных тестах[23].
VP7 — структурный гликопротеин, формирующий наружную поверхность вириона. Он определяет G-тип серологической классификации и вместе с VP4 участвует в формировании иммунитета к инфекции[11].
Неструктурные белки
NSP1, продукт гена 5, является неструктурным РНК-связывающим белком[24]. NSP1 также блокирует продукцию интерферона, части врождённой иммунной системы, защищающей клетки от вирусной инфекции. NSP1 вынуждает протеасомы к лизису ключевых сигнальных компонентов, необходимых для стимуляции продукции интерферона в заражённой клетке и для реакции на интерферон, секретируемый соседними клетками. Целями для протеолитической деградации становятся несколько регуляторных факторов интерферона.[25]
NSP2 — РНК-связывающий белок, аккумулируется в цитоплазматических включениях (вироплазмах) и участвует в репликации генома[26][27].
NSP3 связывается с вирусной мРНК в заражённых клетках и отвечает за выключение синтеза клеточных белков[28]. NSP3 инактивирует два фактора инициации трансляции, необходимые для синтеза белков из мРНК клетки-хозяина. Во-первых, NSP3 выталкивает поли(а)-связывающий белок (PABP) из фактора инициации трансляции eIF4F. PABP необходим для эффективной трансляции транскриптов с 3'-хвостами, которые обнаруживаются у большинства транскриптов клетки-хозяина. Во-вторых, NSP3 инактивирует eIF2, стимулируя его фосфорилирование. В то же время эффективная трансляция вирусной мРНК не требует двух этих факторов, поскольку эта РНК не содержит 3'-концов.[29]
NSP4 — вирусный энтеротоксин, вызывающий диарею. Является первым обнаруженным вирусным энтеротоксином[30].
NSP5 кодируется сегментом 11 генома ротавируса А и в инфицированных вирусом клетках накапливается в вироплазмах[31].
NSP6 является белком, связывающим нуклеиновые кислоты[32], кодируется геном 11 по внефазовой открытой рамке считывания[33].
| Сегмент РНК (Ген) | Размер, спаренных оснований | Белок | Молекулярная масса, kDa | Положение | Копий на частицу | Функция |
|---|---|---|---|---|---|---|
| 1 | 3302 | VP1 | 125 | В вершинах ядра | <25 | РНК-зависимая РНК-полимераза |
| 2 | 2690 | VP2 | 102 | Формирует внутреннюю оболочку ядра | 120 | Стимулирует вирусную РНК-репликазу |
| 3 | 2591 | VP3 | 88 | В вершинах ядра | <25 | метилтрансфераза, мРНК-кэпирующий фермент |
| 4 | 2362 | VP4 | 87 | Шипы на поверхности капсида | 120 | Прикрепление к клетке, вирулентность |
| 5 | 1611 | NSP1 | 59 | Неструктурный белок | 0 | Связывание 5’РНК, блокирование продукции интерферона |
| 6 | 1356 | VP6 | 45 | Толща капсида (средний слой) | 780 | Структурная функция; видоспецифичный антиген |
| 7 | 1104 | NSP3 | 37 | Неструктурный белок | 0 | Усиливает активность вирусной мРНК, выключает синтез клеточных белков |
| 8 | 1059 | NSP2 | 35 | Неструктурный белок | 0 | НТФаза, участвует в упаковке РНК |
| 9 | 1062 | VP71 VP72 | 38 и 34 | На поверхности | 780 | Структурный белок; нейтрализирующий антиген |
| 10 | 751 | NSP4 | 20 | Неструктурный белок | 0 | Энтеротоксин |
| 11 | 667 | NSP5 NSP6 | 22 | Неструктурный белок | 0 | оцРНК- и дцРНК-связывающий модулятор активности NSP2, фосфопротеин |
Таблица составлена на основе штамма обезьяньего ротавируса SA11[34][35][36]. Размеры генов у некоторых других штаммов могут отличаться.
Репликация
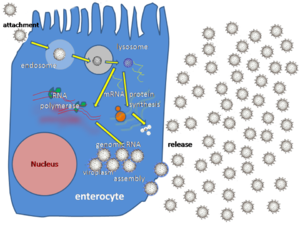
Ротавирусы реплицируются главным образом в кишечнике[37] и заражают энтероциты ворсинок тонкого кишечника, что приводит к структурным и функциональным изменениям эпителия[38]. Тройная белковая оболочка делает их устойчивыми к кислой среде желудка и пищеварительным ферментам в кишечнике.
Существует два возможных пути проникновения вируса в клетку: прямая пенетрация через клеточную мембрану и эндоцитоз. Предполагается, что трансмембранное проникновение опосредовано гидрофобной областью VP5, являющегося продуктом расщепления VP4. Эта область закрыта у нерасщеплённого VP4, поэтому вирионы с белковыми шипами, не подвергшимися расщеплению, не способны проникнуть в клетку этим способом. Второй путь проникновения — эндоцитоз. Вирус проникает в клетку путём опосредованного рецепторами эндоцитоза и образует везикулы, известные как эндосомы. Белки в третьем слое (VP7 и шип VP4) нарушают мембрану эндосомы, создав разницу в концентрации кальция. Это вызывает распад VP7-тримеров на одиночные белковые субъединицы, при этом оставшиеся вокруг вирусной двунитевой РНК белки VP2 и VP6 образуют двухслойную частицу (DLP)[39].
Одиннадцать дцРНК-нитей остаются под защитой двух белковых оболочек, где вирусная РНК-зависимая РНК-полимераза создаёт транскрипты мРНК вирусного генома. Оставаясь в ядре вириона, вирусная РНК избегает врождённого иммунного ответа, называемого РНК-интерференцией и вызываемого присутствием двухцепочечной РНК.
Во время инфекции ротавирус производит мРНК для биосинтеза белка и репликации генов. Большинство ротавирусных белков накапливается в вироплазмах, где реплицируется РНК и собираются DLP. Вироплазмы формируются вокруг ядра клетки уже через два часа после начала вирусной инфекции и состоят из вирусных фабрик, создаваемых, как предполагается, двумя вирусными неструктурными белками: NSP5 и NSP2. Ингибирование NSP5 РНК-интерференцией приводит к резкому снижению репликации ротавирусов. DLP мигрируют в эндоплазматический ретикулум, где они получают свой третий, внешний, слой (образованный VP7 и VP4). Потомство вируса высвобождается из клетки путём лизиса[21][40][41].
См. также
Примечания
- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- 1 2 Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV). (Проверено 26 марта 2017).
- ↑ Атлас по медицинской микробиологии, вирусологии и иммунологии : Учебное пособие для студентов медицинских вузов / Под ред. А. А. Воробьева, А. С. Быкова. — М. : Медицинское информационное агентство, 2003. — С. 117. — ISBN 5-89481-136-8.
- ↑ Грачева Н. М., Аваков А. А., Блохина Т. А., Щербаков И. Т. Клинические аспекты ротавирусной инфекции // Лечащий врач. — 1998. — № 3. — ISSN 1560-5175.
- 1 2 Kirkwood C. D. (September 2010). “Genetic and antigenic diversity of human rotaviruses: potential impact on vaccination programs”. The Journal of Infectious Diseases. 202 (Suppl): S43—S48. DOI:10.1086/653548. PMID 20684716. Используется устаревший параметр
|month=(справка) - ↑ O'Ryan M. (March 2009). “The ever-changing landscape of rotavirus serotypes”. The Pediatric Infectious Disease Journal. 28 (3 Suppl): S60—S62. DOI:10.1097/INF.0b013e3181967c29. PMID 19252426. Используется устаревший параметр
|month=(справка) - ↑ Patton JT (January 2012). “Rotavirus diversity and evolution in the post-vaccine world”. Discovery Medicine. 13 (68): 85—97. PMID 22284787. Используется устаревший параметр
|month=(справка) - ↑ Desselberger U., Wolleswinkel-van den Bosch J., Mrukowicz J., Rodrigo C., Giaquinto C., Vesikari T. (2006). “Rotavirus types in Europe and their significance for vaccination”. Pediatr. Infect. Dis. J. 25 (1 Suppl.): S30—S41. DOI:10.1097/01.inf.0000197707.70835.f3. PMID 16397427.
- ↑ Desselberger, U.; Gray, James. Rotaviruses: methods and protocols / Desselberger, U.; Gray, James. — Totowa, N. J. : Humana Press, 2000. — P. 2. — ISBN 0-89603-736-3.
- ↑ Carter, J., & Saunders, V. A. (2007). Virology: principles and applications. John Wiley & Sons; 148-151
- 1 2 Pesavento J. B., Crawford S. E., Estes M. K., Prasad B. V. (2006). “Rotavirus proteins: structure and assembly”. Curr. Top. Microbiol. Immunol. 309: 189—219. DOI:10.1007/3-540-30773-7_7. PMID 16913048.
- ↑ Prasad B. V., Chiu W. (1994). “Structure of rotavirus”. Curr. Top. Microbiol. Immunol. 185: 9—29. PMID 8050286.
- ↑ Patton J. T. (1995). “Structure and function of the rotavirus RNA-binding proteins”. J. Gen. Virol. 76 (11): 2633—2644. DOI:10.1099/0022-1317-76-11-2633. PMID 7595370. Архивировано из оригинала (PDF) 2012-12-09. Проверено 2013-03-28.
- ↑ Patton J. T. (2001). “Rotavirus RNA replication and gene expression”. Novartis Found. Symp. 238: 64—77, discussion 77—81. DOI:10.1002/0470846534.ch5. PMID 11444036.
- ↑ Vásquez-del Carpió R., Morales J. L., Barro M., Ricardo A., Spencer E. (2006). “Bioinformatic prediction of polymerase elements in the rotavirus VP1 protein”. Biol. Res. 39 (4): 649—659. DOI:10.4067/S0716-97602006000500008. PMID 17657346.
- ↑ Arnoldi F., Campagna M., Eichwald C., Desselberger U., Burrone O. R. (2007). “Interaction of rotavirus polymerase VP1 with nonstructural protein NSP5 is stronger than that with NSP2”. J. Virol. 81 (5): 2128—2137. DOI:10.1128/JVI.01494-06. PMC 1865955. PMID 17182692.
- ↑ Angel J., Franco M. A., Greenberg H. B. Desk Encyclopedia of Human and Medical Virology / Mahy B. W. J., Van Regenmortel M. H. V. — Boston : Academic Press, 2009. — P. 277. — ISBN 0-12-375147-0.
- ↑ Cowling V. H. (January 2010). “Regulation of mRNA cap methylation”. Biochem. J. 425 (2): 295—302. DOI:10.1042/BJ20091352. PMC 2825737. PMID 20025612. Используется устаревший параметр
|month=(справка) - ↑ Gardet A., Breton M., Fontanges P., Trugnan G., Chwetzoff S. (2006). “Rotavirus spike protein VP4 binds to and remodels actin bundles of the epithelial brush border into actin bodies”. J. Virol. 80 (8): 3947—3956. DOI:10.1128/JVI.80.8.3947-3956.2006. PMC 1440440. PMID 16571811.
- ↑ Arias C. F., Isa P., Guerrero C. A., Méndez E., Zárate S., López T., Espinosa R., Romero P., López S. (2002). “Molecular biology of rotavirus cell entry”. Arch. Med. Res. 33 (4): 356—361. DOI:10.1016/S0188-4409(02)00374-0. PMID 12234525.
- 1 2 Jayaram H., Estes M. K., Prasad B. V. (April 2004). “Emerging themes in rotavirus cell entry, genome organization, transcription and replication”. Virus Research. 101 (1): 67—81. DOI:10.1016/j.virusres.2003.12.007. PMID 15010218. Используется устаревший параметр
|month=(справка) - ↑ Bishop R. F. (1996). “Natural history of human rotavirus infection”. Arch. Virol. Suppl. 12: 119—128. PMID 9015109.
- ↑ Beards G. M., Campbell A. D., Cottrell N. R., Peiris J. S., Rees N., Sanders R. C., Shirley J. A., Wood H. C., Flewett T. H. (1 February 1984). “Enzyme-linked immunosorbent assays based on polyclonal and monoclonal antibodies for rotavirus detection” (PDF). J. Clin. Microbiol. 19 (2): 248—254. PMC 271031. PMID 6321549.
- ↑ Hua J., Mansell E. A., Patton J. T. (1993). “Comparative analysis of the rotavirus NS53 gene: conservation of basic and cysteine-rich regions in the protein and possible stem-loop structures in the RNA”. Virology. 196 (1): 372—378. DOI:10.1006/viro.1993.1492. PMID 8395125.
- ↑ Arnold MM (2016). “The Rotavirus Interferon Antagonist NSP1: Many Targets, Many Questions”. Journal of Virology. 90 (11): 5212—5. DOI:10.1128/JVI.03068-15. PMID 27009959.
- ↑ Kattoura M. D., Chen X., Patton J. T. (1994). “The rotavirus RNA-binding protein NS35 (NSP2) forms 10S multimers and interacts with the viral RNA polymerase”. Virology. 202 (2): 803—813. DOI:10.1006/viro.1994.1402. PMID 8030243.
- ↑ Taraporewala Z. F., Patton J. T. (2004). “Nonstructural proteins involved in genome packaging and replication of rotaviruses and other members of the Reoviridae”. Virus Res. 101 (1): 57—66. DOI:10.1016/j.virusres.2003.12.006. PMID 15010217.
- ↑ Poncet D., Aponte C., Cohen J. (1 June 1993). “Rotavirus protein NSP3 (NS34) is bound to the 3' end consensus sequence of viral mRNAs in infected cells” (PDF). J. Virol. 67 (6): 3159—3165. PMC 237654. PMID 8388495.
- ↑ López, S; Arias, CF (August 2012). “Rotavirus-host cell interactions: an arms race”. Current Opinion in Virology. 2 (4): 389—98. DOI:10.1016/j.coviro.2012.05.001. PMID 22658208.
- ↑ Hyser J. M., Estes M. K. (January 2009). “Rotavirus vaccines and pathogenesis: 2008”. Current Opinion in Gastroenterology. 25 (1): 36—43. DOI:10.1097/MOG.0b013e328317c897. PMC 2673536. PMID 19114772. Используется устаревший параметр
|month=(справка) - ↑ Afrikanova I., Miozzo M. C., Giambiagi S., Burrone O. (1996). “Phosphorylation generates different forms of rotavirus NSP5”. J. Gen. Virol. 77 (9): 2059—2065. DOI:10.1099/0022-1317-77-9-2059. PMID 8811003. Архивировано из оригинала 2012-05-26. Проверено 2013-04-18.
- ↑ Rainsford E. W., McCrae M. A. (2007). “Characterization of the NSP6 protein product of rotavirus gene 11”. Virus Res. 130 (1—2): 193—201. DOI:10.1016/j.virusres.2007.06.011. PMID 17658646.
- ↑ Mohan K. V., Atreya C. D. (2001). “Nucleotide sequence analysis of rotavirus gene 11 from two tissue culture-adapted ATCC strains, RRV and Wa”. Virus Genes. 23 (3): 321—329. DOI:10.1023/A:1012577407824. PMID 11778700.
- ↑ Desselberger U. Rotavirus: basic facts. In Rotaviruses Methods and Protocols. Ed. Gray, J. and Desselberger U. Humana Press, 2000, pp. 1—8. ISBN 0-89603-736-3
- ↑ Patton J. T. Rotavirus RNA replication and gene expression. In Novartis Foundation. Gastroenteritis Viruses, Humana Press, 2001, pp. 64—81. ISBN 0-471-49663-4
- ↑ Claude M. Fauquet; J. Maniloff; Desselberger, U. Virus taxonomy: classification and nomenclature of viruses: 8th report of the International Committee on Taxonomy of Viruses. — Amsterdam : Elsevier/Academic Press, 2005. — P. 489. — ISBN 0-12-249951-4.
- ↑ Greenberg H. B., Estes M. K. (May 2009). “Rotaviruses: from pathogenesis to vaccination”. Gastroenterology. 136 (6): 1939—1951. DOI:10.1053/j.gastro.2009.02.076. PMID 19457420. Используется устаревший параметр
|month=(справка) - ↑ Greenberg H. B., Clark H. F., Offit P. A. (1994). “Rotavirus pathology and pathophysiology”. Curr. Top. Microbiol. Immunol. 185: 255—283. PMID 8050281.
- ↑ Baker M., Prasad B. V. (2010). “Rotavirus cell entry”. Current Topics in Microbiology and Immunology. 343: 121—148. DOI:10.1007/82_2010_34. PMID 20397068.
- ↑ Patton J. T., Vasquez-Del Carpio R., Spencer E. (2004). “Replication and transcription of the rotavirus genome”. Curr. Pharm. Des. 10 (30): 3769—3777. DOI:10.2174/1381612043382620. PMID 15579070.
- ↑ Ruiz M. C., Leon T., Diaz Y., Michelangeli F. (2009). “Molecular biology of rotavirus entry and replication”. TheScientificWorldJournal. 9: 1476—1497. DOI:10.1100/tsw.2009.158. PMID 20024520.
Данная страница на сайте WikiSort.ru содержит текст со страницы сайта "Википедия".
Если Вы хотите её отредактировать, то можете сделать это на странице редактирования в Википедии.
Если сделанные Вами правки не будут кем-нибудь удалены, то через несколько дней они появятся на сайте WikiSort.ru .